Abstract
For obtaining accurate and reliable gene expression results it is essential that quantitative realtime RT-PCR (qRT-PCR) data are normalized with appropriate reference genes. The current exponential increase in postgenomic studies on the honey bee, Apis mellifera, makes the standardization of qRT-PCR results an important task for ongoing community efforts. For this aim we selected four candidate reference genes (actin, ribosomal protein 49, elongation factor 1-alpha, tbp-association factor) and used three software-based approaches (geNorm, BestKeeper and NormFinder) to evaluate the suitability of these genes as endogenous controls. Their expression was examined during honey bee development, in different tissues, and after juvenile hormone exposure. Furthermore, the importance of choosing an appropriate reference gene was investigated for two developmentally regulated target genes. The results led us to consider all four candidate genes as suitable genes for normalization in A. mellifera. However, each condition evaluated in this study revealed a specific set of genes as the most appropriated ones.
Zusammenfassung
Die quantitative real-time RT-PCR (qRT-PCR) Methode enwickelt sich zu einer der am häufigsten benutzten Methode zur Quantifizierung von mRNAs. Für exakte und zuverlässig reproduzierbare Ergebnisse ist es allerdings notwendig, dass die jeweiligen qRT-PCR Werte mittels eines geeigneten Referenzgens normalisiert werden. Trotz der Verfügbarkeit von Referenzgen-Studien bei verschiedenen Organismen ist die Bewertung geeigneter Referenzgene für den jeweiligen Organismus, in diesem Fall die Honigbiene Apis mellifera, erforderlich. Genau dies war das Ziel der vorliegenden Untersuchung, und dafür wählten wir folgende Gene als Kandidaten aus: actin, rp49, ef1-alpha and tbp-af (Tab. I). Diese wurden mittels der Computerprogramme geNorm, BestKeeper und Normfinder auf ihre Eignung und Eigenschaften als Referenzgene getestet. Wir untersuchten dies in verschiedenen biologischen Zusammenhängen: in verschiedenen Phasen der postembryonalen Entwicklung, in verschiedenen Geweben und Organen und nach Behandlung mit Juvenilhormon (JH-III).
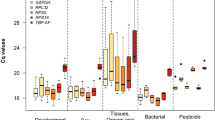
Für Studien zur Postembryonalentwicklung erwiesen sich bei Verwendung von geNorm actin und tbp-af und bei Verwendung von BestKeeper und NormFinder rp49 als die jeweils stabilsten Gene (Tab. II und III). Trotz dieser Unterschiede in der Rangfolge der Eignung zeigten alle vier Referenzgene für das Entwicklungsstadium F3 die höchsten mRNA-Werte an (Abb. 1). Dies weist darauf hin, dass die Regulierung der Genexpression in der Entwicklung in weitem Maße korreliert ist, und dass sie in der Larval- und Pupalentwicklung wahrscheinlich stark von physiologischen Faktoren beeinflusst wird. Ähnliche Unterschiede in der Eignung als Referenzgene waren auch bei den Gewebestudien und nach JH-III-Behandlung zu sehen. Während geNorm in diesen Situationen actin und ef1-alpha als die am besten geeigneten Gene auswies (Tab. II und III), waren dies rp49 und ef1-alpha, wenn BestKeeper und NormFinder als Bewertungsprogramme eingesetzt wurden (Tab. II und III). Obwohl bei den Vergleichen verschiedener Gewebe und Organe die Expressionswerte aller vier Gene signifikant schwankten (Abb. 2), wurden sie bei den BestKeeper- und geNorm-Analysen als stabile Referenz-Gene bewertet. Für Untersuchungen zu Behandlungen mit JH-III waren die stabilsten Gene ef1-alpha und tbp-af (geNorm), tbp-af (Best-Keeper) und actin (NormFinder) (Tab. II and III). Da wir für die Kontrollen (Aceton behandelteArbeiterinnen) und die JH-III behandelten Arbeiterinnen keine Unterschiede in der Expression dieser Gene fanden (Abb. 3), zeigt dies, dass bei solchen Versuchen alle vier Gene zur Normalisierung eingesetzt werden können.
In einem weiteren Versuchsansatz untersuchten wir, welche Auswirkungen die Auswahl von Referenzgenen auf die jeweiligen Zielgene hat. Für die Zielgene jhe-like und proPO fanden wir, dass die Normalisierung mittels eines weniger geeigneten Referenzgens eine deutliche Auswirkung auf die Berechnung der relativen Genexpressionswwerte hat, insbesondere, wenn die Proben niedrige mRNA-Werte haben (Abb. 4).
Unsere Untersuchung zeigt, dass alle vier Gene im Prinzip als Referenzgene für quantitative Genexpressionsstudien bei A. mellifera eingesetzt werden können. Da jedoch jeder biologische Kontext eine jeweils andere Kombination von Referenzgenen als am besten geeignet auswies, schlagen wir vor, dass jeweils immer zwei dieser Gene für die Normalisierung verwendet werden.
Similar content being viewed by others
References
Andersen C.L., Jensen J.L., Orntoft T.F. (2004) Normalization of real-time quantitative reverse transcription-PCR data: a model-based variance estimation approach to identify genes suited for normalization, applied to bladder and colon cancer data sets, Cancer Res. 64, 5245–5250.
Bitondi M.M., Nascimento A.M., Cunha A.D., Guidugli K.R., Nunes R.M., Simões Z.L. (2006) Characterization and expression of the Hex 110 gene encoding a glutamine-rich hexamerin in the honey bee, Apis mellifera, Arch. Insect Biochem. Physiol. 63, 57–72.
Bustin S.A. (2002) Quantification of mRNA using real-time reverse transcription PCR (RT-PCR): trends and problems, J. Mol. Endocrinol. 29, 23–39.
Bustin S.A., Benes V., Nolan T., Pfaffl M.W. (2005) Quantitative real-time RT-PCR-a perspective, J. Mol. Endocrinol. 34, 597–601.
Corona M., Velarde R.A., Remolina S., Moran-Lauter A., Wang Y., Hughes K.A., Robinson G.E. (2007) Vitellogenin, juvenile hormone, insulin signaling, and queen honey bee longevity, Proc. Natl. Acad. Sci. USA 104, 7128–7133.
de Kok J.B., Roelofs R.W., Giesendorf B.A., Pennings J.L., Waas E.T., Feuth T., Swinkels, D.W., Span P.N. (2005) Normalization of gene expression measurements in tumor tissues: comparison of 13 endogenous control genes, Lab. Invest. 85, 154–159.
Evans J.D. (2004) Transcriptional immune responses by honey bee larvae during invasion by the bacterial pathogen, Paenibacillus larvae, J. Invertebr. Pathol. 85, 105–111.
Evans J.D. (2006) Beepath: an ordered quantitative-PCR array for exploring honey bee immunity and disease, J. Invertebr. Pathol. 93, 135–139.
Evans J.D., Wheeler D.E. (2000) Expression profiles during honeybee caste determination, Genome Biol. 2, research0001.1-0001.6.
Grozinger C.M., Sharabash N.M., Whitfield C.W., Robinson G.E. (2003) Pheromone-mediated gene expression in the honey bee brain, Proc. Natl. Acad. Sci. USA 100, 14519–14525.
Huggett J., Dheda K., Bustin S., Zumla A. (2005) Realtime RT-PCR normalisation; strategies and considerations, Genes Immun. 6, 279–284.
Judice C.C., Carazzole M.F., Festa F., Sogayar M.C., Hartfelder K., Pereira G.A. (2006) Gene expression profiles underlying alternative caste phenotypes in a highly eusocial bee, Melipona quadrifasciata, Insect Mol. Biol. 15, 33–44.
Kucharski R., Maleszka R. (2005) Microarray and real-time PCR analyses of gene expression in the honeybee brain following caffeine treatment, J. Mol. Neurosci. 27, 269–276.
Lee P.D., Sladek R., Greenwood C.M., Hudson T.J. (2002) Control genes and variability: absence of ubiquitous reference transcripts in diverse mammalian expression studies, Genome Res. 12, 292–297.
Lourenço A.P., Zufelato M.S., Bitondi M.M., Simões Z.L. (2005) Molecular characterization of a cDNA encoding prophenoloxidase and its expression in Apis mellifera, Insect Biochem. Mol. Biol. 35, 541–552.
Mackert A.S., Nascimento A.M., Bitondi M.M.G., Hartfelder K., Simões Z.L. (2008) Identification of a juvenile hormone esterase-like in the honey bee, Apis mellifera L.-expression analysis and functional assays, Comp. Biochem. Physiol. B. doi: 10.1016/j.cbpb.2008.01.004.
Pfaffl M.W., Tichopad A., Prgomet C., Neuvians T.P. (2004) Determination of stable housekeeping genes, differentially regulated target genes and sample integrity: BestKeeper—Excel-based tool using pair-wise correlations, Biotechnol. Lett. 26, 509–515.
Rachinsky A., Strambi C., Strambi A., Hartfelder K. (1990) Caste and metamorphosis: hemolymph titers of juvenile hormone and ecdysteroids in last instar honeybee larvae, Gen. Comp. Endocrinol. 79, 31–38.
Ririe K.M., Rasmussen R.P., Wittwer C.T. (1997) Product differentiation by analysis of DNA melting curves during the polymerase chain reaction, Anal. Biochem. 245, 154–160.
Rozen S., Skaletsky H. (2000) Primer3 on the WWW for general users and for biologist programmers, Methods Mol. Biol. 132, 365–386.
Schmittgen T.D., Zakrajsek B.A. (2000) Effect of experimental treatment on housekeeping gene expression: validation by real-time, quantitative RT-PCR, J. Biochem. Biophys. Methods 46, 69–81.
Shimokawa T., Kato M., Ezaki O., Hashimoto S. (1998) Transcriptional regulation of musclespecific genes during myoblast differentiation, Biochem. Biophys. Res. Commun. 246, 287–292.
Silver N., Best S., Jiang J., Thein S.L. (2006) Selection of housekeeping genes for gene expression studies in human reticulocytes using real-time PCR, BMC Mol. Biol. 7, 33.
Suzuki T., Higgins P.J., Crawford D.R. (2000) Control selection for RNA quantitation, Biotechniques 29, 332–337.
Szabo A., Perou C.M., Karaca M., Perreard L., Quackenbush J.F., Bernard P.S. (2004) Statistical modeling for selecting housekeeper genes, Genome Biol. 5, research59.
The Honey Bee Genome Sequencing Consortium (2006) Insights into social insects from the genome of the honey bee Apis mellifera, Nature 443, 931–949.
Thellin O., Zorzi W., Lakaye B., De Borman B., Coumans B., Hennen G., Grisar T., Igout A., Heinen E. (1999) Housekeeping genes as internal standards: use and limits, J. Biotechnol. 75, 291–295.
Vandesompele J., De Preter K., Pattyn F., Poppe B., Van Roy N., De Paepe A., Speleman F. (2002) Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes, Genome Biol. 3, research0034.
Wheeler D.E., Buck N., Evans J.D. (2006) Expression of insulin pathway genes during the period of caste determination in the honey bee, Apis mellifera, Insect Mol. Biol. 15, 597–602.
Whitfield C.W., Cziko A.M., Robinson G.E. (2003) Gene expression profiles in the brain predict behavior in individual honey bees, Science 302, 296–299.
Willems E., Mateizel I., Kemp C., Cauffman G., Sermon K., Leyns L. (2006) Selection of reference genes in mouse embryos and in differentiating human and mouse ES cells, Int. J. Dev. Biol. 50, 627–635.
Yamazaki Y., Shirai K., Paul R.K., Fujiyuki T., Wakamoto A., Takeuchi H., Kubo T. (2006) Differential expression of HR38 in the mushroom bodies of the honeybee brain depends on the caste and division of labor, FEBS Lett. 580, 2667–2670.
Zou Z., Lopez D.L., Kanost M.R., Evans J.D., Jiang H. (2006) Comparative analysis of serine protease-related genes in the honey bee genome: possible involvement in embryonic development and innate immunity, Insect Mol. Biol. 15, 603–614.
Author information
Authors and Affiliations
Corresponding author
Additional information
Manuscript editor: Marla Spivak
Rights and permissions
About this article
Cite this article
Lourenço, A.P., Mackert, A., Cristino, A.d.S. et al. Validation of reference genes for gene expression studies in the honey bee, Apis mellifera, by quantitative real-time RT-PCR. Apidologie 39, 372–385 (2008). https://doi.org/10.1051/apido:2008015
Received:
Revised:
Accepted:
Issue Date:
DOI: https://doi.org/10.1051/apido:2008015