Abstract
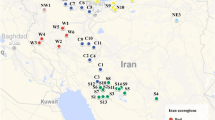
Crop improvement is an important approach to overcome challenges raised from future uncertainties of agricultural systems and growing human population. The fundamental need for such improvement efforts is the availability of well characterized plant germplasm with sufficient genetic diversity. The 2C DNA is defined as the nuclear DNA content of an unreplicated diploid cell (in G1 phase) and is used both to get an estimate of genome size and ploidy level. Flow cytometry provides accurate and fast estimation of the genome size of plants. The genus Avena belongs to Poaceae (Gramineae) family and includes approximately 30 species including common oat (A. sativa). There are 837 Avena accessions in the USDA (United States Department of Agriculture) Germplasm Resources Information Network (USDA-GRIN) collected from Turkey. This also is the largest ex situ Avena collection from Turkey. However, initial genomic characterization of the collection has not yet been conducted. We estimated genome sizes and determined ploidy levels of Turkish oat collection. Nuclear DNA content of accessions ranged from 25.66 to 26.56 pg for A. sativa, from 25.48 to 26.88 pg for A. sterilis and from 24.85 to 26.41 pg for A. fatua. The average and range values for all three hexaploid species were in a similar close range. The range for putative tetraploid accessions belonging to A. barbata was from 12.79 to 16.90 pg. We found a number of aggregates. Furthermore, there was a negative correlation found between altitude and genome size. Obtained results will help to better utilize Avena collection in breeding efforts.
Zusammenfassung
Die Verbesserung von Nutzpflanzen ist ein wichtiger Ansatz, um die Herausforderungen zu bewältigen, die sich aus den zukünftigen Unsicherheiten der landwirtschaftlichen Systeme und der wachsenden Bevölkerung ergeben. Die Grundvoraussetzung für solche Verbesserungsbemühungen ist die Verfügbarkeit von gut charakterisiertem Keimgewebe der Pflanze mit ausreichender genetischer Vielfalt. Die 2C-DNA ist definiert als der Kern-DNA-Gehalt einer nicht replizierten diploiden Zelle (in der G1-Phase) und wird sowohl zur Abschätzung der Genomgröße als auch des Ploidiegrads verwendet. Die Durchflusszytometrie ermöglicht eine genaue und schnelle Abschätzung der Genomgröße von Pflanzen. Die Gattung Avena gehört zur Familie der Poaceae (Gramineae) und umfasst etwa 30 Arten, darunter auch den gemeinen Hafer (A. sativa). Im USDA (United States Department of Agriculture) Germplasm Resources Information Network (USDA-GRIN) wurden 837 Avena-Akzessionen aus der Türkei gesammelt. Dies ist gleichzeitig die größte ex-situ-Avena-Sammlung aus der Türkei. Eine erste genomische Charakterisierung der Sammlung ist jedoch noch nicht erfolgt. Wir schätzten die Genomgrößen und ermittelten die Ploidiegrade der türkischen Hafersammlung. Der Kern-DNA-Gehalt lag bei A. sativa zwischen 25,66 und 26,56 pg, bei A. sterilis zwischen 25,48 und 26,88 pg und bei A. fatua zwischen 24,85 und 26,41 pg. Die Werte für alle drei hexaploiden Arten lagen in einem ähnlichen Bereich. Die Werte für mutmaßlich tetraploide Akzessionen von A. barbata lagen zwischen 12,79 und 16,90 pg. Wir haben eine Reihe von Aggregaten gefunden. Außerdem wurde eine negative Korrelation zwischen Höhenlage und Genomgröße festgestellt. Die erzielten Ergebnisse werden dazu beitragen, die Avena-Sammlung besser für Züchtungsbemühungen nutzen zu können.

Similar content being viewed by others
References
Bennett MD (1972) Nuclear DNA content and minimum generation time in herbaceous plants. Proc R Soc Lond, B, Biol Sci 181:109
Brummer EC, Cazcarro PM, Luth D (1999) Ploidy determination of alfalfa germplasm accessions using flow cytometry. Crop Sci 39:1202–1207
Doležel J, Bartos J (2005) Plant DNA flow cytometry and estimation of nuclear genome size. Ann Bot 95:99–110
Doležel J, Binarová P, Lucretti S (1989) Analysis of nuclear DNA content in plant cells by flow cytometry. Biol Plant 31:113–120
Greilhuber J (2005) Intraspecific variation in genome size in angiosperms: identifying its existence. Ann Bot 95:91–98
Grime JP, Shacklock JML, Band SR (1985) Nuclear-DNA contents, shoot phenology and species co-existence in a limestone grassland community. New Phytol 100:435–445
Harlan JR, de Wet JM (1971) Toward a rational classification of cultivated plants. Taxon 20:509–517
Havananda T, Brummer EC, Maureira-Butler IJ, Doyle JJ (2010) Relationships among diploid members of the (Fabaceae) species complex based on chloroplast and mitochondrial DNA sequences. Syst Botany 35(1):140–150
Macgillivray CW, Grime JP (1995) Genome size predicts frost-resistance in British herbaceous plants—implications for rates of vegetation response to global warming. Funct Ecol 9:320–325
Rasane P, Jha A, Sabikhi L, Kumar A, Unnikrishnan VS (2015) Nutritional advantages of oats and opportunities for its processing as value added foods—a review. J Food Sci Technol 52:662–675
Reeves G, Francis D, Davies MS, Rogers HJ, Hodkinson TR (1998) Genome size is negatively correlated with altitude in natural populations of Dactylis glomerate. Ann Bot 82:99–105
Sakiroglu M, Brummer EC (2011) Clarifying the ploidy of some accessions in the USDA alfalfa germplasm collection. Turk J Bot 35:509–519
Sakiroglu M, Brummer EC (2013) Presence of phylogeographic structure among wild diploid alfalfa accessions (Medicago sativa L. subsp microcarpa Urb.) with evidence of the center of origin. Genet Resour Crop Evol 60:23–31
Sakiroglu M, Kaya MM (2012) Estimating genome size and confirming ploidy levels of wild tetraploid alfalfa accessions (Medicago sativa subsp x varia) using flow cytometry. Turk J Field Crop 17:151–156
Sakiroglu M, Doyle JJ, Brummer EC (2010) Inferring population structure and genetic diversity of broad range of wild diploid alfalfa (Medicago sativa L.) accessions using SSR markers. Theor Appl Genet 121:403–415
Swift H (1950) The constancy of desoxyribose nucleic acid in plant nuclei. Proc Nat Acad Sci 36(11):643–654
Tuna M, Vogel KP, Arumuganathan K, Gill KS (2001) DNA content and ploidy determination of bromegrass germplasm accessions by flow cytometry. Crop Sci 41:1629–1634
Yan H, Martin SL, Bekele WA, Latta RG, Diederichsen A, Peng Y, Tinker NA (2016) Genome size variation in the genus Avena. Genome 59(3):209–220
Acknowledgements
Authors thank Gulru Yucel and Taha Tangut for technical assistance.
Author information
Authors and Affiliations
Corresponding author
Ethics declarations
Conflict of interest
M.A. Akbudak, M. Şakiroğlu and M. Tuna declare that they have no competing interests.
Rights and permissions
About this article
Cite this article
Akbudak, M.A., Şakiroğlu, M. & Tuna, M. Estimation of Nuclear DNA Content and Determination of Relationship Between Altitude and Genome Size of USDA Turkish Oat (Avena spp.) Collection. Gesunde Pflanzen 70, 171–178 (2018). https://doi.org/10.1007/s10343-018-0428-x
Received:
Accepted:
Published:
Issue Date:
DOI: https://doi.org/10.1007/s10343-018-0428-x